R语言学习
R源码安装常见报错及更换package安装源
统计检验应用
卡方检验案例应用
linux本地安装ggplot2
shell脚本中运行R代码
conda构建虚拟环境
karyoploteR基因组数据可视化
Linux环境中R语言绘图问题
【实用】Bioconductor的正确使用
本文档使用 MrDoc 发布
-
+
up
down
首页
karyoploteR基因组数据可视化
2023-07-19 10:59:17 星期三 ## karyoploteR介绍 > ikaryoploteR is an R package to create karyoplots, that is, representations of whole genomes with arbitrary data plotted on them. It is inspired by the R base graphics system and does not depend on other graphics packages. The aim of karyoploteR is to offer the user an easy way to plot data along the genome to get broad genome-wide view to facilitate the identification of genome wide relations and distributions. karyoploteR,一个适用于所有基因组数据(any data on any genome)非圆环布局(non-circular layouts)的可视化 R/Bioconductor 包。开发该包的团队的目标是致力于打造一款能够像 Circos 那样灵活多变,但又可以满足用户更方便地使用直线(straight lines)而不是圆环(circles)展示基因组数据的工具。 ## 教程 Bioconductor地址: http://bioconductor.org/packages/karyoploteR/ Github地址: https://github.com/bernatgel/karyoploter 文献: https://doi.org/10.1093/bioinformatics/btx346 案例教程:https://bernatgel.github.io/karyoploter_tutorial/ ## 安装 ```if (!require("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("karyoploteR") ``` ## 实战测试 ``` ## 导入相关依赖包 library(ggplot2) library(karyoploteR) library(biomaRt) ## 设置参数及读取相关数据 setwd("E:/4.生信流程/5.CNVseq/CNVseq相关文件/报告手绘出图/10163/") sample="10163" file1="10163.cnvall.txt" ## 读取文件并设置数据格式 data1 <- read.table(file = file1, sep="\t", header=TRUE, stringsAsFactors = FALSE, blank.lines.skip=TRUE, skipNul=TRUE) print(names(data1)) data1<-data1[,c("Chr","Start","End","Ratio")] names(data1)<-c('chr','start','end','ratio') data1$ratio<-(data1$ratio)*2 data1$pos<-(data1$start+data1$end)/2 if(nrow(data1[which(data1$ratio>4),])>1){ data1[which(data1$ratio>4),]$ratio<-4 } ## 转换成染色体区间 dataA <- toGRanges(data1[,c('chr', 'pos','pos','ratio')]) ## 绘制无间隙的染色体图 # 输出到 png outname<-paste(sample,'-WES-CNV-all.png',sep='') png(outname,width=1800,height=300, units = "px") pp <- getDefaultPlotParams(plot.type=4) # 查看默认参数设置 pp$ideogramlateralmargin <- 0 # 修改某一个参数,两个染色体中间就没有了缝隙 #kp <- plotKaryotype(genome="hg19", plot.type=4, cytobands = GRanges(), labels.plotter = NULL, plot.params=pp, main=paste(sample," CNV plot",sep='')) kp <- plotKaryotype(genome="hg19", plot.type=4, cytobands = GRanges(), labels.plotter = NULL, plot.params=pp) #kpDataBackground(kp, col=c("#E1EAF2", NA), r0=-0.05, r1=0.95) # 先加背景色,否则会压住轴线 kpDataBackground(kp, col=c(NA), r0=-0.05, r1=0.95) #kpPoints(kp, data=dataA, ymax = 4, ymin=0, col="#00B0F0", y=dataA$ratio, r0=-0.05, r1=0.95, cex = 0.2) kpAxis(kp, ymax = 4, ymin=0, numticks=5, label.margin = 0.01, r0=-0.05, r1=0.95, cex=0.8) # 添加轴 kpAddChromosomeNames(kp, chr.names=c(1:22, 'X', 'Y'), cex.lab=2, lwd=2) # 加染色体名 kpAddLabels(kp, labels="Copy Number", pos=3, label.margin = 0.03, srt=90, lwd=2) kpAddChromosomeSeparators(kp, col="#666666", lty=3) chros<-unique(data1$chr) colsa<-c("#00B0F0", "grey") n = 0 # 按染色体一条条绘制 for(chro in chros){ n<-n+1 dataTmp<-data1[which(data1$chr==chro),] dataTmp <- toGRanges(dataTmp[,c('chr', 'pos','pos','ratio')]) kpPoints(kp, data=dataTmp, ymax = 4, ymin=0, col=colsa[n%%2+1], y=dataTmp$ratio, r0=-0.05, r1=0.95, cex = 0.2) #kpPoints(kp, chr="chr15", x=23684689, y=3, ymax = 4, ymin=0, cex=.5, r0=-0.05, r1=0.95, col="red") #添加单点,那就可以对CNV部分的点进行其他颜色设置了 } #kpAbline(kp, h=c(2), ymax = 4, ymin=0,col="black", lwd=0.5, r0=-0.05, r1=0.95) #添加水平线 #kpAbline(kp, h=c(1, 3), ymax = 4, ymin=0,col="red", lwd=0.5, r0=-0.05, r1=0.95) #添加水平线 kpAbline(kp, h=1, chr="chrX", ymax = 4, ymin=0,col="red", lwd=2, r0=-0.05, r1=0.95) #添加水平线 #kpAbline(kp, h=3, chr="chr15", ymax = 4, ymin=0,col="blue", lwd=2, r0=-0.05, r1=0.95) #添加重复水平线 kpSegments(kp, chr="chr15", x0=23684689, x1=28557995, y0=3, y1=3, ymax = 4, ymin=0, lwd=4, r0=-0.05, r1=0.95, col="red") #添加某一个片段 dev.off() ## 绘制单条染色体的细节 ############ for chrX plot ############# outname<-paste(sample,'-WES-CNV-one.png',sep='') png(outname,width=1800,height=300, units = "px") chro<-"chrX" kp <- plotKaryotype(plot.type = 2, chromosomes = chro) dataTmp<-data1[which(data1$chr==chro),] dataTmp <- toGRanges(dataTmp[,c('chr', 'pos','pos','ratio')]) kpAxis(kp,r0=-0.05, r1=0.95, data.panel=1, ymin=0, ymax=4, side = 1, numticks = 5, tick.pos = c(1, 3), labels = c('loss', 'gain')) kpPoints(kp, data=dataTmp, y=dataTmp$ratio, col=colsa[1], r0=-0.05, r1=0.95, ymin=0, ymax=4, cex = 0.2) kpAbline(kp, h=1, r0=-0.05, r1=0.95, col='red', ymin=0, ymax=4, lty=2, cex=0.1) #Loss kpAbline(kp, h=3, r0=-0.05, r1=0.95, col='red', ymin=0, ymax=4, lty=2, cex=0.1) #Gain kpSegments(kp, chr="chrX", x0=23684689, x1=28557995, y0=3, y1=3, ymax = 4, ymin=0, lwd=4, r0=-0.05, r1=0.95, col="red") #添加某一个片段 dev.off() ################################################################################## ``` 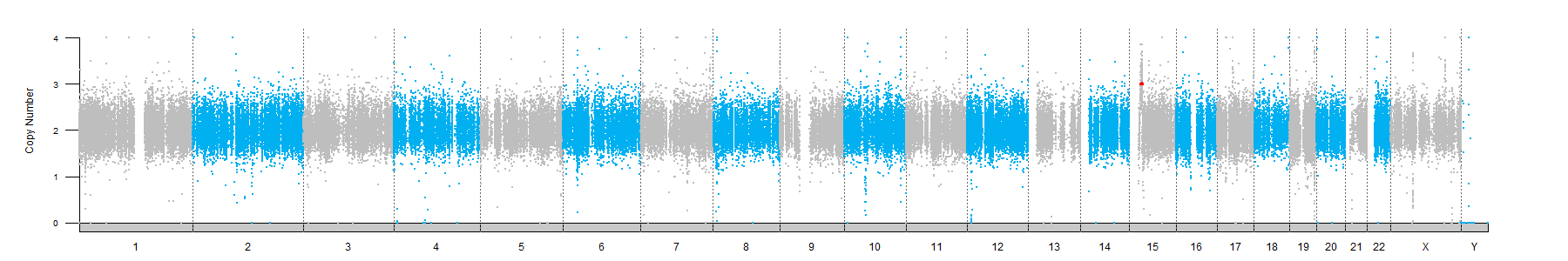 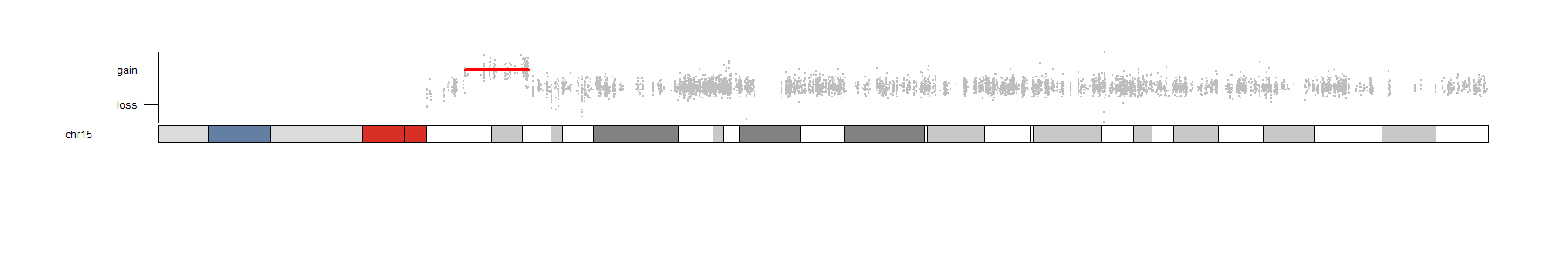
laihui126
2024年7月31日 09:27
分享文档
收藏文档
上一篇
下一篇
微信扫一扫
复制链接
手机扫一扫进行分享
复制链接
关于 MrDoc
觅道文档MrDoc
是
州的先生
开发并开源的在线文档系统,其适合作为个人和小型团队的云笔记、文档和知识库管理工具。
如果觅道文档给你或你的团队带来了帮助,欢迎对作者进行一些打赏捐助,这将有力支持作者持续投入精力更新和维护觅道文档,感谢你的捐助!
>>>捐助鸣谢列表
微信
支付宝
QQ
PayPal
下载Markdown文件
分享
链接
类型
密码
更新密码