Tumor
癌症序列变异解释和报告的标准和指南(ASCO和CAP联合推荐)
肺癌分析插件Can_28修复问题
Chom化疗插件及bug修复
Brca插件
FACTERA-fusionGene
SEGF-挖掘NGS中融合基因的新方法
maftools-肿瘤突变数据可视化神器
从数据库中获取免疫相关基因(IRGs)
数据库10KIP—基于ImmPort免疫组学数据挖掘
TCGA数据分析
下载分析TCGA数据库的数据
差异表达分析(limma & edgeR & DESeq2)
TCGA数据的规律【更新中】
生存分析
生存模型构建
突变数据
本文档使用 MrDoc 发布
-
+
up
down
首页
maftools-肿瘤突变数据可视化神器
## 一、安装maftools 根据maftools官网:https://bioconductor.org/packages/release/bioc/vignettes/maftools/inst/doc/maftools.html ,按如下命令安装: ``` if (!require("BiocManager")) install.packages("BiocManager") BiocManager::install("maftools") ``` 但是发现,安装失败,有许多依赖包会提示不支持R-3.5版本,解决方法: 对于需要BiocManager进行安装的包(如GenomeInfoDb,BiocParallel),使用如下安装 ``` #R交互界面中 library('BiocManager') BiocManager::install("pkg_name") ``` 而常规的包,如“RCurl”可以通过: ``` #R交互界面中 install.packages('RCurl') #或者在CRAN上下载好源码,如wordcloud,mclust等(https://cran.r-project.org/web/packages/) 通过如下安装: R CMD INSTALL mclust_5.4.2.tar.gz ``` ## 二、使用简介 假设你已经全面学习了肿瘤外显子测序数据的分析,得到了你所有配对样本的somatic mutation信息,而且制作成了还算标准的maf格式文件,那么就可以跟着这篇教程进行可视化啦。 全部流程在R里面运行,maftools提供了直接读取maf文件的接口,而且存储为S4对象,非常方便进行一系列可视化操作。 而且大部分都是一行代码出图,基本上不需要太高深的R语言知识,现学现用。 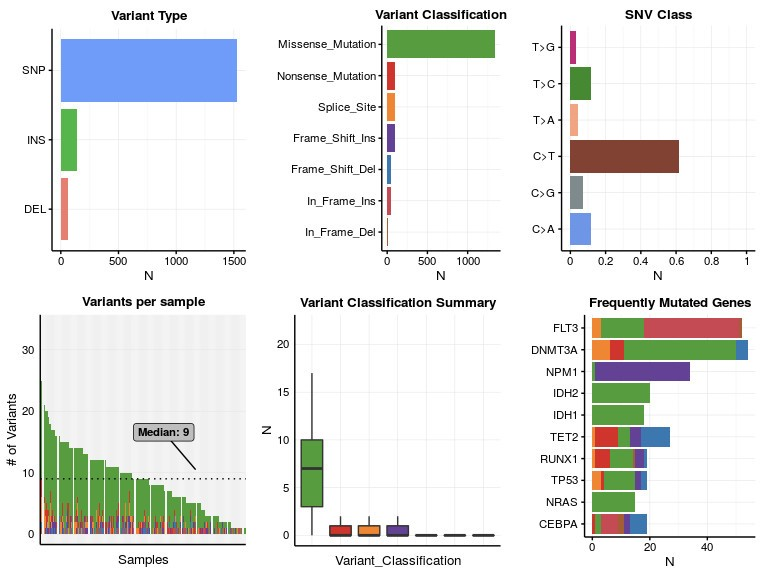 而且得到zuguan大神的complexHeatmap的oncoplot也被封装成了一行代码: oncoplot(maf = laml, top = 10, removeNonMutated = TRUE) 即可出图如下:  还有以前我在生信菜鸟团博客介绍过的,MutationMapper on cBioPortal 的lollipopPlot  最近比较火的TMB也囊括进来了: [下载TCGA所有癌症的maf文件计算TMB](http://mp.weixin.qq.com/s?__biz=MzAxMDkxODM1Ng==&mid=2247484947&idx=1&sn=fc4873fd0dbdf8c5d6431c2d42017151&chksm=9b4846a8ac3fcfbe091ffcafa3deea5e2c66823309012b3cd8d5cdca1c970e222d102b1c8a9d&scene=21#wechat_redirect) 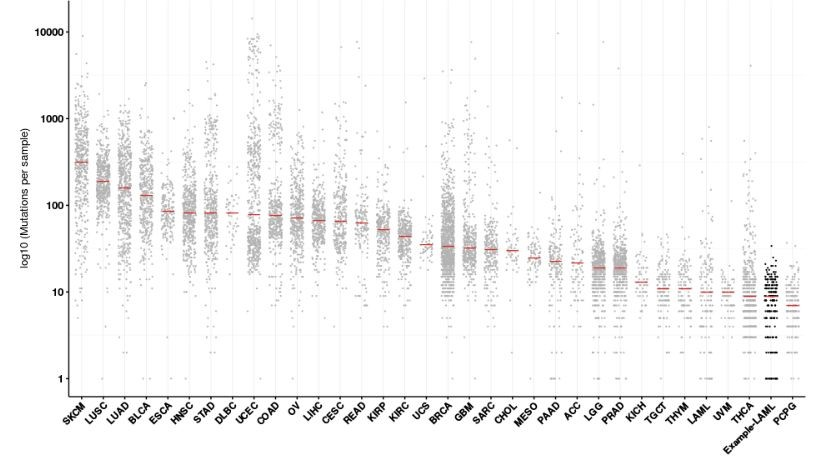 华而不实的词云也少不了咯:  还有探索MAF分布:  其实图并不重要,重要的是理解图背后的生物学医院。 最重要的是: 马上就可以下载TCGA的maf文件,玩一下这个包吧,肯定对你有帮助的!!
laihui126
2023年1月9日 15:17
分享文档
收藏文档
上一篇
下一篇
微信扫一扫
复制链接
手机扫一扫进行分享
复制链接
关于 MrDoc
觅道文档MrDoc
是
州的先生
开发并开源的在线文档系统,其适合作为个人和小型团队的云笔记、文档和知识库管理工具。
如果觅道文档给你或你的团队带来了帮助,欢迎对作者进行一些打赏捐助,这将有力支持作者持续投入精力更新和维护觅道文档,感谢你的捐助!
>>>捐助鸣谢列表
微信
支付宝
QQ
PayPal
下载Markdown文件
分享
链接
类型
密码
更新密码